Résumé
Les gastroentérites virales sont principalement dues aux rotavirus et aux norovirus. Les rotavirus sont l’étiologie majeure des gastroentérites aiguës de l’enfant. La surveillance moléculaire des génotypes de rotavirus en France et en Europe a montré une variabilité temporelle et géographique. Les souches G1 et G9 étaient prédominantes suivies des G2, G3 et G4. Toutes ces souches étaient majoritairement associées à P [8] donnant une certaine stabilité aux combinaisons génotypiques qui peut assurer une efficacité du vaccin à court et moyen terme. Néanmoins, la présence de souches atypiques G12 et G8, susceptibles d’émerger dans l’avenir, nécessite de poursuivre la surveillance de ces virus. Les norovirus appartiennent à la famille des Caliciviridae. Ces virus à ARN présentent une grande diversité, on distingue cinq génogroupes divisés eux-mêmes en génotypes. A cette diversité s’ajoute une évolutivité génétique par recombinaison et surtout par mutation. De nouvelles souches sont responsables des vagues épidémiques régulièrement constatées.
Summary
Rotaviruses and noroviruses are the main causes of acute gastroenteritis in young children and adults, respectively. Prospective molecular surveillance of rotavirus genotypes in France and Europe shows that circulating strains may vary with the season, locality or country. Rotavirus OK? genotypes G1 and G9 are the most prevalent. Most strains are associated with P[8], showing a certain genotypic stability of rotaviruses currently circulating in Europe and suggesting that vaccination would be effective at least in the short and medium term. However, atypical strains G12 and G8 must be monitored in case they emerge in future. Noroviruses belong to the Caliciviridae family. These single-stranded RNA viruses show major genetic diversity: they are divided into 5 genogroups, which are themselves subdivided into genotypes. In addition, new variants are continually arising, and are frequently associated with new epidemic waves of gastroenteritis.
INTRODUCTION
Les gastroentérites infectieuses sont une des premières causes de morbidité et de mortalité à travers le monde : chaque année on dénombre près de 1,4 milliard d’épisodes et 1,6 à 2,5 millions de décès, majoritairement dans les pays en voie de développement [1]. Les virus en sont les principales étiologies et parmi ceux-ci les rotavirus et les norovirus prédominent. Les rotavirus étant plus fréquemment retrouvés chez l’enfant de moins de cinq ans alors que les norovirus sont l’agent majeur des gastroentérites de l’adulte notamment des personnes âgées en institution.
LES ROTAVIRUS
Les rotavirus, décrits pour la première fois en 1973 par Bishop et al . [2], sont classés dans la famille des
Reoviridae . Cette famille comprend des virus non-enveloppés, résistants à l’inactivation physique, dont le génome est un ARN double brin segmenté (figure 1). Les rotavirus comportent sept groupes antigéniques distincts (A à G). Seuls les rotavirus des groupes A, B et C peuvent infecter l’homme. Ceux du groupe A sont de loin les plus importants puisqu’ils sont responsables des gastroentérites aiguës chez l’enfant en bas âge. Les groupes B et C ne sont responsables que d’épidémies limitées ou d’infections sporadiques.
Transmission-épidémiologie
La transmission des rotavirus est avant tout féco-orale, interhumaine, par contacts directs ou indirects par les surfaces et objets contaminés par les fèces ou les vomissures. La résistance des rotavirus et leur dose infectieuse faible facilitent leur diffusion. La voie aéroportée paraît également possible mais ne constitue pas une voie ordinaire de contamination.
Diversité des rotavirus
Au sein du groupe A, la classification des rotavirus est relativement complexe et repose sur les caractéristiques antigéniques des deux protéines de capside externe :
VP7 définissant les antigènes G (pour glycoprotein ) et VP4 définissant les antigènes
P (pour protease-sensitive ). A ce jour, on distingue 14 sérotypes G et 14 sérotypes P.
Néanmoins, pour des raisons de disponibilité et de spécificité des réactifs immuno-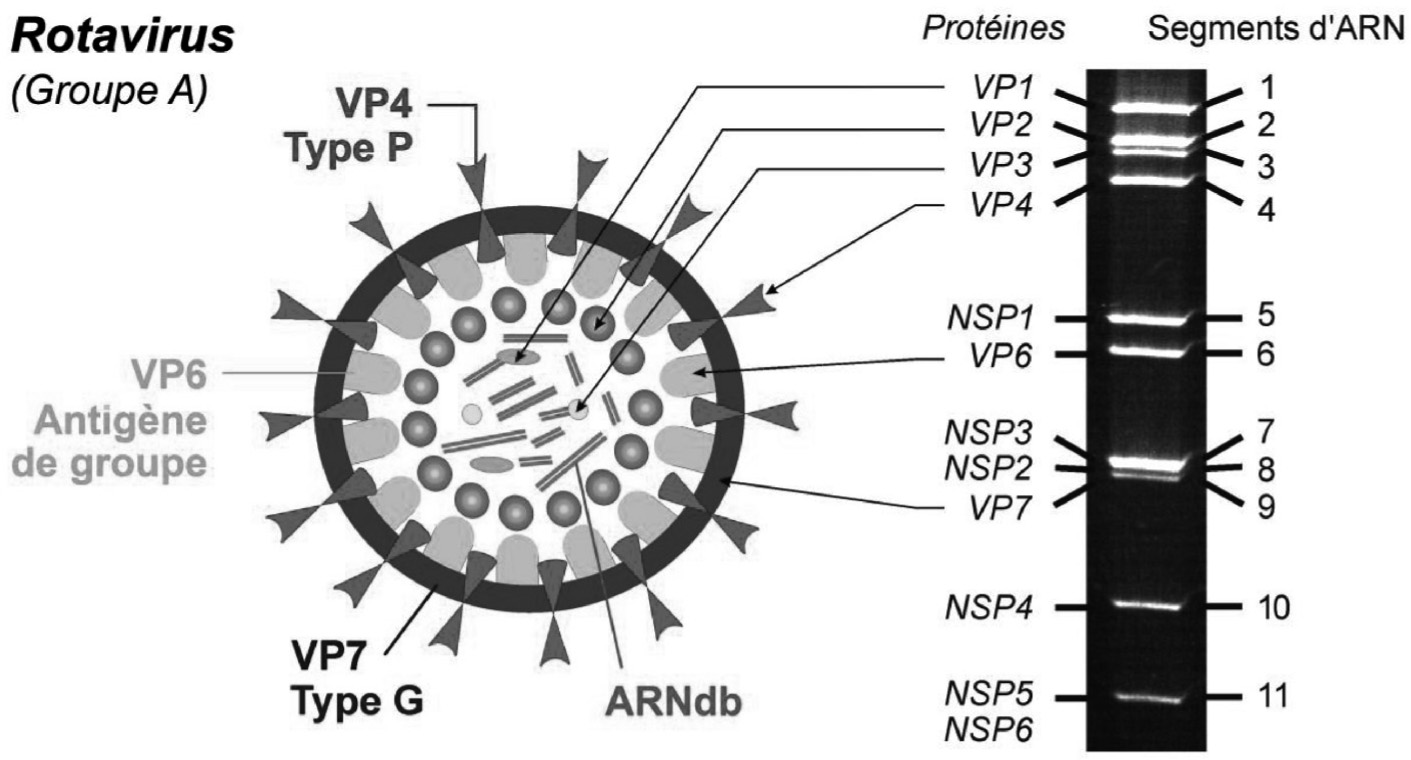
Fig. 1. — Organisation du génome et structure des rotavirus logiques le génotypage des ARN codant ces deux protéines leur est préféré. Ainsi, pour les rotavirus du groupe A, 15 génotypes G et 27 génotypes P ont pu être définis, dont 11 G et 15 P ont été identifiés chez des souches de rotavirus ayant été à l’origine d’une gastroentérite chez l’homme.
De cette classification génétique et antigénique à deux valences résulte une très grande diversité des combinaisons de souches de rotavirus. Néanmoins, les diverses études épidémiologiques conduites à travers le monde montrent que cinq combinaisons génotypiques sont responsables à elles seules de 90 % des infections à rotavirus chez l’homme [3] : les souches G1P [8], G2P [4], G3P [8], G4P [8] et G9P [8]. Des génotypes G inhabituels tels que G5, G6, G8 G10 ou G12 émergent dans quelques régions du monde, notamment dans les régions tropicales. Elles sont pour certaines le résultat d’une transmission inter-espèce de rotavirus animaux — bovins, porcins pour les plus fréquents — qui ont pu passer à l’homme et à l’origine de réassortiments homme-animal.
Les souches G1P [8] sont les plus répandues dans le monde. Cette prédominance des G1P [8] semblerait due à l’émergence de nouvelles souches plutôt qu’à la réémergence de souches anciennes. La persistance de la circulation des souches G1 pourrait s’expliquer par le fait que des variants antigéniques (G1a, G1b…) pourraient apparaître et disparaître sous l’influence de la pression immunitaire [4]. Les variations des autres génotypes se manifestent sous forme d’épidémies revenant par périodes de deux ou trois saisons consécutives.
En France, la surveillance ininterrompue des souches de rotavirus ces dix dernières années a confirmé que les souches G1P [8] constituaient la combinaison génotypique la plus commune suivies des souches G9P [8] avec respectivement une prévalence moyenne de 59,2 % et 25 % pour les trois dernières saisons de l’étude [5]. La prévalence des autres génotypes était plus faible, les infections mixtes représentaient 3 % des isolats et étaient dominées par les combinaisons G1/G9. Au contraire de la diversité des génotypes G, la répartition des génotypes P montre une nette prédominance des souches P [8] avec des taux de détection moyens de 92,9 %.
Avec une faible prévalence, plusieurs souches recombinantes ou atypiques ont été détectées au cours de l’étude. Les rotavirus G12, généralement associés avec le génotype P [8] d’origine humaine, représentaient les souches atypiques les plus fréquentes et ont été détectées dans six villes différentes au cours des trois saisons.
Tous les G12 possédaient des gènes NSP4 et VP6 d’origine humaine. Des souches G8 ont été également détectées. Les souches G8P [6], régulièrement détectées à Paris au cours de l’étude, et la souche G8P [14] possédaient des gènes NSP4 et VP6 d’origine bovine. L’analyse phylogénétique du gène VP7 a montré que les souches parisiennes G8P [6] appartenaient à une lignée différente de la souche G8P[14], cette dernière possédait 99,4 % d’homologie nucléotidique avec une souche bovine isolée dans la région de Dijon. Ainsi les souches G8P [6] semblaient relativement adaptées à l’Homme et capables d’une transmission interhumaine au contraire de la souche G8P [14] dont le passage à l’Homme semblait récent. Quelques souches G6 d’origine animale ont également été détectées. Les autres souches détectées étaient des combinaisons G/P inhabituelles de génotypes communs d’origine humaine animale tels que P [6] ou encore P [3], P [9] et P [14].
Variabilité temporelle et géographique [5]
Les faits le plus marquants de cette étude ont été de montrer la variabilité des génotypes de rotavirus selon les années et les villes étudiées et l’émergence d’un génotype nouveau de rotavirus. Le génotype G9P [8] rarement détecté jusqu’en 2003-2004 est brusquement apparu comme la principale combinaison génotypique de rotavirus avec environ 60 % des souches de la saison 2004-2005 alors que le génotype G1P [8] en représentaient moins de 10 %. Depuis, les souches G9P [8] se maintiennent, en France, à un niveau d’incidence relativement stable d’environ 25 % chaque année et représentent le deuxième type commun le plus fréquent (figure 2).
L’émergence de cette souche durant la saison 2004-2005 a été observée dans tous les pays européens, mais à l’inverse de la France, la prévalence globale de ce génotype a baissé dans la plupart des pays européens et il ne représentait plus que 8 % des souches détectées en Europe contre 45,8 % pour le génotype G1P [8, 6].
Les autres génotypes ont également montré des variations saisonnières, mais moins marquées. Pour les souches G2P [4], les variations constatées (2,0 % à 13,6 %) reflètent la diversité intersaisonnière normale des rotavirus, entraînée par la réémergence des antigènes VP7 mutés. Ceci devrait être surveillé soigneusement au cours du programme de vaccination français lors des prochaines saisons puisque l’un des vaccins commercialisés n’inclut pas cette combinaison génotypique dans sa préparation.
* Les données de 2001 à 2006 sont issues des trois CHU préfigurant la mise en place du réseau national (Paris Saint-Vincent de Paul, Limoges et Dijon).
Fig. 2. — Évolution des combinaisons génotypiques de rotavirus en France entre 2001 et 2009* et distribution cumulée des combinaisons génotypiques entre 2006 et 2009.
La répartition des génotypes G de rotavirus par ville et par année montre qu’il existe des circulations hétérogènes des deux souches dominantes selon la saison et la ville (figure 3). La prévalence des souches G9P [8] était épisodiquement plus importante que celle des souches G1P [8] pour sept villes selon la saison, décrivant ainsi un mouvement relatif vers le sud-est. Les infections à rotavirus ont été détectées tout au long de l’année avec un pic entre janvier et mars. L’analyse des données des cinq villes les plus informatives a montré des variations temporelles dans l’apparition de ce pic d’infections en fonction de la saison et du lieu. Dans les villes de province, les pics d’épidémies hivernales sont survenus deux mois plus tard, entre février et mars, qu’à Paris où le pic s’est produit entre janvier et février. Les données de la surveillance européenne confirment cette variabilité temporelle et géographique des génotypes de rotavirus, chaque pays ayant son propre profil de prévalence des génotypes de rotavirus [6].
AT : Hôpital Armand-Trousseau ; RD : Hôpital Robert-Debré ; SVP : Hôpital Saint-Vincent de Paul.
Fig. 3. — Répartition temporelle et géographique des génotypes de rotavirus entre 2006 et 2009 en France métropolitaine Risque d’émergence de nouvelles souches
La survenue d’infections par des virus d’origine potentiellement animale a été régulièrement rapportée dans la littérature. Dans la surveillance que nous avons conduite en France ces dix dernières années, les souches de génotype G6 et G8, qui possédaient aussi des gènes NSP4 et VP6 d’origine bovine, semblaient pour certaines adaptées à l’Homme. Mais leur aptitude actuelle semble insuffisante pour permettre une transmission interhumaine facile.
Cependant, nous devons rester prudents et l’apparition récente des rotavirus de génotype G9 doit rester en mémoire. Ce génotype, également retrouvé chez le porc et le mouton, a été détecté pour la première fois chez des enfants diarrhéiques à Philadelphie aux USA en 1983. Il a disparu durant une décennie et réapparaît en 1995 dans cette même ville et dans tous les USA. Mais il reste peu fréquent ; des cas sporadiques sont détectés en Europe, Afrique et Asie jusqu’à la fin des années 90. à partir des années 2000 il devient rapidement l’un des principaux génotypes en Australie et en Europe durant la saison 2004-2005. L’analyse phylogénétique des souches montre trois linéages différents apparus successivement : les souches de lignée phylogénétique une, isolées aux USA et au Japon durant les années 1980, les souches de lignée deux, isolées durant la même période mais seulement en Inde et les souches de la lignée trois, isolées à partir de 1993-1994 et correspondant aux souches récentes responsables de l’émergence de ce génotype dans l’ensemble du globe. Ce génotype s’est modifié par mutations successives et par réassortiments sélectifs avec d’autres segments génomiques pour finalement s’adapter parfaitement à l’homme.
LES NOROVIRUS
Les norovirus ont été identifiés en 1972 par microscopie électronique dans un échantillon de selle provenant d’un malade contaminé au cours d’une épidémie survenue dans une école a Norwalk (Ohio, États-Unis) en 1968 [7]. D’abord dénommés « agents de Norwalk » en référence à la ville de leur première identification, ils sont aujourd’hui regroupés dans le genre Norovirus et classés dans la famille des
Caliciviridae . Ces petits virus, non enveloppés, ont un génome constitué d’un
ARN simple brin positif polyadénylé comprenant trois cadres ouverts de lecture (ORF1-3, open reading frame ) codant respectivement les protéines non structurales, la protéine majeure de capside (VP1) et une protéine structurale mineure basique (VP2). La protéine VP1 est divisé en deux domaines : S à la base de la protéine et à l’intérieur de la particule et P plus externe. Ce domaine P est lui-même subdivisé en deux régions P1et P2. La région P2, hypervariable, est située à l’extérieur de la particule (figure 4) ; elle joue un rôle majeur dans l’attachement au(x) récepteur(s) et dans la réponse immune humorale.
Mode de transmission
Le mode féco-oral est la principale voie de transmission du virus, soit de façon directe, soit par la contamination des surfaces et de l’environnement. Les aérosols lors des vomissements peuvent représenter un mode de transmission opportuniste.
La résistance du virus, la dose infectieuse faible et la présence de porteurs asymptomatiques ou une excrétion prolongée facilitent la diffusion du virus. La transmission par les aliments ou l’eau des infections à norovirus est bien connue, mais c’est la transmission de personne à personne qui est le plus souvent incriminée dans les gastroentérites hivernales ou les épidémies dans les établissements pour personnes âgées.
Pathogenèse et susceptibilité de l’hôte
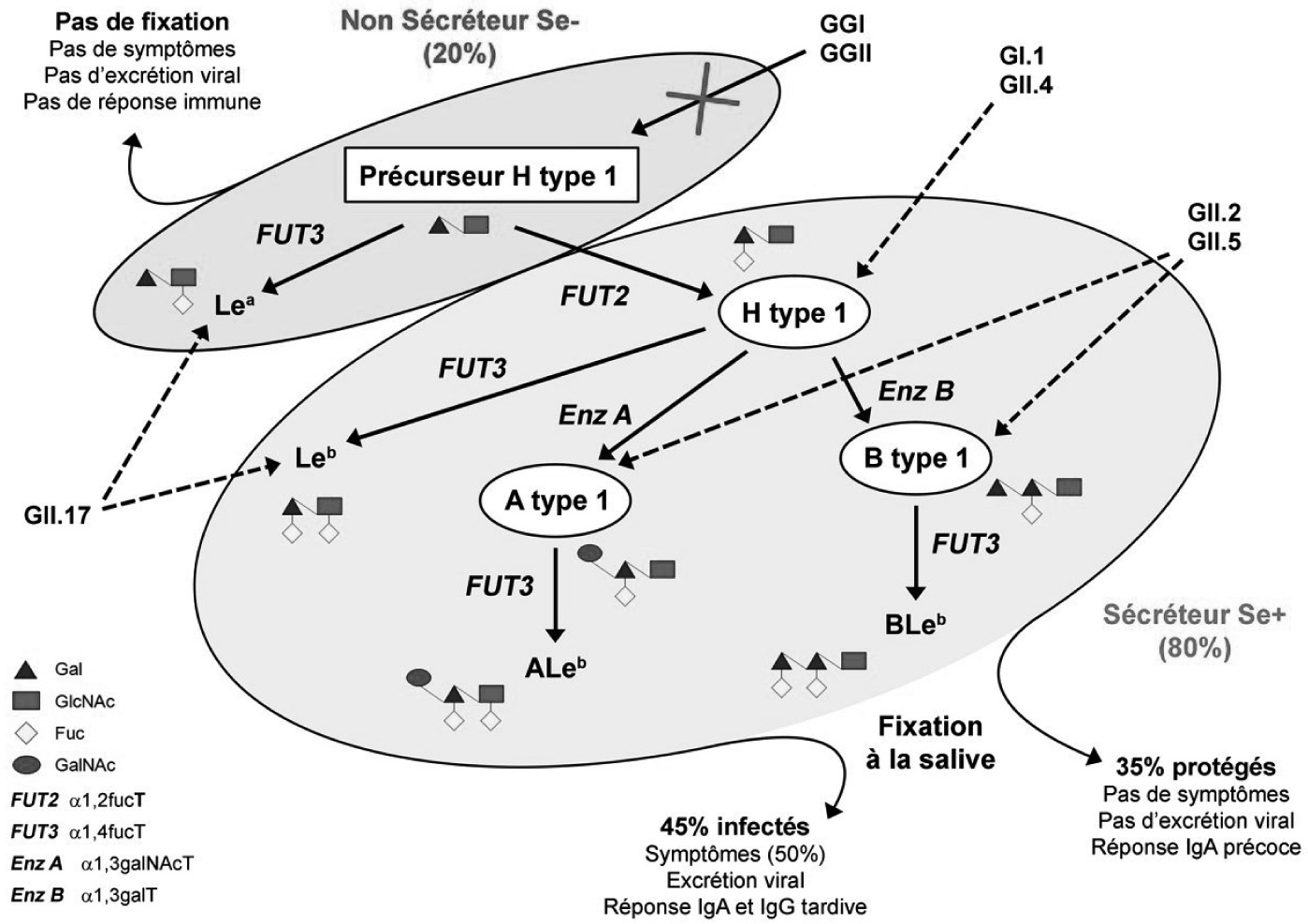
Les premières expérimentations chez les volontaires sains montraient que malgré une bonne réponse immunologique après infection, la protection induite était de courte durée. Mais curieusement, ces études montraient aussi que 13 a 40 % des volontaires n’étaient jamais infectés et seulement 50 % d’entre eux devenaient symptomatiques. À l’origine inexpliquée, cette différence de susceptibilité est due à des facteurs génétiques de l’hôte et plus particulièrement à la présence ou non de récepteurs glycannes au niveau des cellules intestinales [8, 9]. Ces récepteurs glycannes appartiennent à la famille des groupes sanguins tissulaires ABO dont
Fig. 4. — Organisation du génome et structure des norovirus l’expression dépend du polymorphisme combiné aux locus ABO , FUT2 ( Fucosyltransferase 2) et FUT3 (Fucosyltransferase 3, groupe sanguin Lewis) . Les différentes souches de norovirus ne reconnaissant pas le même motif glycanne [10], cela explique les variations individuelles observées lors des expérimentations ou lors des épidémies (Figure 5).
Diversité génétique et évolution des norovirus
Les norovirus présentent une grande diversité génétique. Les souches sont classées en génogroupes subdivisés en génotypes. Actuellement, on distingue cinq génogroupes (I a V) mais seuls les génogroupes I, II et IV infectent l’homme. Les génogroupes I et II sont les plus importants et sont divisés respectivement en 8 et 19 génotypes [11]. Ainsi la dénomination utilisée mentionne le génogroupe puis le génotype du norovirus : GGI.1 a GGI.8 et GGII.1 a GGII.19. Certains norovirus sont aussi responsables de zoonoses : des souches porcines appartenant au génogroupe II, des souches bovines regroupées dans le génogroupe III, et une souche murine ou MNV (génogroupe V). Ce MNV est actuellement le seul norovirus qui peut être mis en culture et donc être utilisé dans des modèles animaux pour l’étude des norovirus humains. Contrairement aux rotavirus, il n’existe aujourd’hui aucune preuve de transmission des norovirus de l’animal à l’homme même si certaines souches présentent des similitudes génétiques.
Les norovirus sont l’objet d’une variabilité génétique résultant de recombinaisons ou de mutations. Les recombinaisons entre des souches exprimant une ARN polymérase et une capside dérivées de souches parentales différentes ont été décrites.
L’un de ces virus recombinants, le variant GGIIb, a été isolé pour la première fois au cours d’une épidémie d’origine hydrique dans le sud de la France en août 2000 [12].
Fig. 5. — Interactions des norovirus humains avec leurs ligands glycannes et conséquences pour l’expression symptomatique de la maladie. Les interactions des norovirus humains avec leurs ligands sont indiquées par des flèches pointillées. Les motifs glucidiques constituant chaque ligand sont représentés symboliquement. Les enzymes permettant la synthèse des glycannes sont indiqués sur des flèches pleines. La population se divise en deux groupes, les non sécréteurs (20 % de la population) et les sécréteurs, ces derniers affichant les glycannes nécessaires à la fixation des norovirus sur la muqueuse intestinale. Sauf exceptions (GGII.17), les individus non sécréteurs ne pouvant fixer les norovirus ne contractent pas de gastroentérites et la réponse immunitaire est absente. Le groupe des sécréteurs se divise en deux sous-groupes. Pour 45 % de la population totale, les patients développent la maladie, excrètent le virus et présentent une élévation tardive des IgA (immunoglobulines A) et IgG spécifiques. Pour l’autre groupe, soit 35 % de la population, les personnes sont asymptomatiques, n’excrètent pas de virus et ont, en revanche, une réponse IgA précoce.
Il a représenté jusqu’à 26 % des souches caractérisées en Europe et reste fréquemment retrouvé lors d’épidémies et de cas sporadiques de gastroentérite. D’autres virus recombinants ont été identifiés suggérant que ce phénomène n’est pas rare.
La variabilité génétique par mutation concerne tous les norovirus mais plus particulièrement le génotype GGII.4, principal responsable des épidémies à travers le monde. L’analyse génétique des souches successives durant la dernière décennie montre la grande capacité évolutive de ce génotype et l’émergence de variants à l’origine de nouvelles épidémies à diffusion mondiale. Ce fut le cas en 2002 avec la souche Farmington Hills entraînant une augmentation importante des épidémies dans toute l’Europe [13], en 2004 avec la souche
Hunter qui lui a succédé et en 2006 avec la circulation simultanée de deux nouveaux variants, les souches 2006a et surtout les souches 2006b [14, 15] (Figures 6 et 7). Ces souches nouvelles sont porteuses de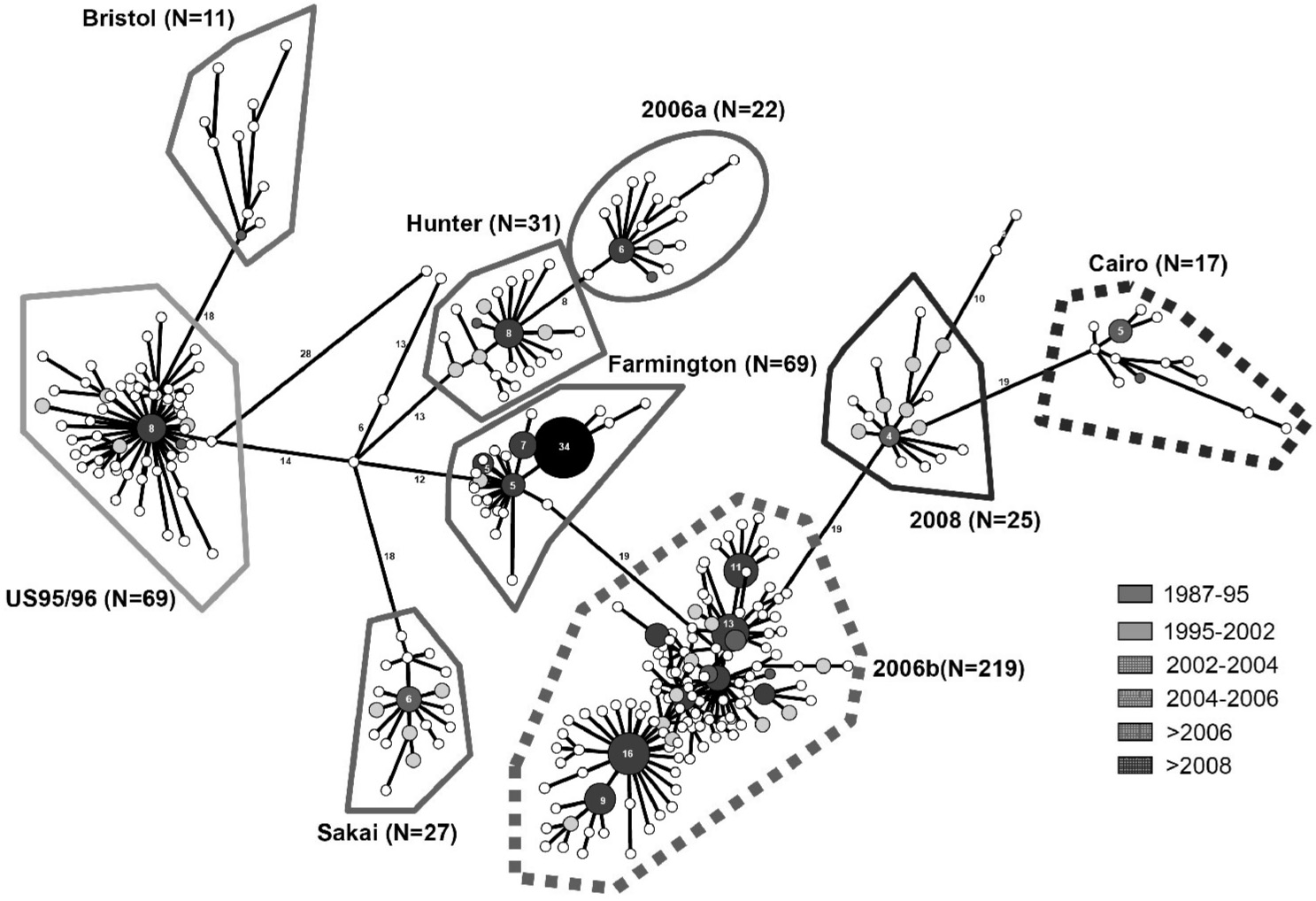
Fig. 6. — Évolution des épidémies de gastroentérites dues aux différents variants de norovirus en Europe entre 2000 et 2008.
Fig. 7. — Schéma de la filiation des différentes souches de norovirus mutations dans la protéine de capside VP1 (région P2) qui peuvent être à l’origine d’un échappement à la réponse immunitaire ou d’une modification des propriétés de liaison de ces virus à leurs récepteurs cellulaires [15].
CONCLUSIONS
Les rotavirus et le norovirus, principaux virus des gastroentérites, présentent une grande diversité et évolutivité génétique. Ces phénomènes peuvent faciliter leur diffusion dans la population malgré une pression immunitaire induite par les précédentes infections. Néanmoins, les mécanismes semblent différents pour ces deux virus : essentiellement liés à des réassortiments génétiques pour les rotavirus et à des mutations pour les norovirus. La nécessité d’une parfaite adaptation à l’Homme des différents segments génomiques du rotavirus et la relative constance du génotype P[8] au sein des différentes combinaisons génotypiques responsables de la grandes majorité des infections laissent des espoirs quant à l’efficacité à long terme des vaccins anti-rotavirus. Mais pour ces deux virus, il est nécessaire de poursuivre la surveillance épidémiologique des souches virales circulant dans la population.
BIBLIOGRAPHIE [1] Bryce J., Boschi-Pinto C., Shibuya K., Black R.E. — (2005) WHO estimates of the causes of death in children. Lancet, 365, (9465), 1147-1152.
[2] Bishop R.F., Davidson G.P., Holmes I.H., Ruck B.J. — (1973) Virus particles in epithelial cells of duodenal mucosa from children with acute non-bacterial gastroenteritis. Lancet 2, (7841), 1281-1283.
[3] Santos N., Hoshino Y. — (2005) Global distribution of rotavirus serotypes/genotypes and its implication for the development and implementation of an effective rotavirus vaccine. Rev. Med.
Virol., 15 (1), 29-56.
[4] Arista S., Giammanco G.M., De Grazia S., Ramirez S., Lo Biundo C., Colomba C., Cascio A., Martella V. — (2006) Heterogeneity and temporal dynamics of evolution of G1 human rotaviruses in a settled population. J. Virol., 80 (21), 10724-10733.
[5] De Rougemont A., Kaplon J., Pillet S., Mory O., Gagneur A., Minoui-Tran A., Meritet J.F., Mollat C., Lorrot M., Foulongne V., Gillet Y., Nguyen-Bourgain C., Alain S., Agius G., Lazrek M., Colimon R., Fontana C., Gendrel D., Pothier P. — Molecular and Clinical Characterization of Rotavirus From Diarrheal Infants Admitted to Pediatric Emergency Units in France. Pediatr. Infect. Dis. J.
[6] Iturriza-Gomara M., Dallman T., Banyai K., Bottiger B., Buesa J., Diedrich S., Fiore L., Johansen K., Koopmans M., Korsun N., Koukou D., Kroneman A., Laszlo B., Lappalainen M., Maunula L., Marques A.M., Matthijnssens J., Midgley S., Mladenova Z., Nawaz S., Poljsak-Prijatelj M., Pothier P., Ruggeri F.M., Sanchez-Fauquier A., Steyer A., Sidaraviciute-Ivaskeviciene I., Syriopoulou V., Tran A.N., Usonis V., Vanr M., Der A., Gray J. — Rotavirus genotypes co-circulating in Europe between 2006 and 2009 as determined by EuroRotaNet, a pan-European collaborative strain surveillance network.
Epidemiol. Infect., 1-15.
[7] Kapikian A.Z. — (2000) The discovery of the 27-nm Norwalk virus: an historic perspective.
J.
Infect. Dis ., 181, Suppl. 2, S295-302.
[8] Marionneau S., Ruvoen N., Le Moullac-Vaidye B., Clement M., Cailleau-Thomas A., Ruiz-Palacois G., Huang P., Jiang X., Le Pendu J. — (2002) Norwalk virus binds to histo-blood group antigens present on gastroduodenal epithelial cells of secretor individuals .
Gastroenterology, 122 (7), 1967-1977.
[9] Lindesmith L., Moe C., Marionneau S., Ruvoen N., Jiang X., Linblad L., Stewart P., Lependu J., Baric R. — (2003) Human susceptibility and resistance to Norwalk virus infection. Nat. Med., 9 (5), 548-553.
[10] Harrington P.R., Lindesmith L., Yount B., Moe C.L., Baric R.S. — (2002) Binding of Norwalk virus-like particles to ABH histo-blood group antigens is blocked by antisera from infected human volunteers or experimentally vaccinated mice. J. Virol., 76 , (23), 12335-12343.
[11] Zheng D.P., Ando T., Fankhauser R.L., Beard R.S., Glass R.I., Monroe S.S. — (2006) Norovirus classification and proposed strain nomenclature. Virology, 346 (2), 312-323.
[12] Ambert-Balay K., Bon F., Le Guyader F., Pothier P., Kohli E. — (2005) Characterization of new recombinant noroviruses. J. Clin. Microbiol ., 43 (10), 5179-5186.
[13] Lopman B., Vennema H., Kohli E., Pothier P., Sanchez A., Negredo A., Buesa J., Schreier E., Reacher M., Brown D., Gray J., Iturriza M., Gallimore C., Bottiger B., Hedlund K.O., Torven M., Von Bonsdorff C.H., Maunula L., Poljsak-Prijatelj M., Zimsek J., Reuter G., Szucs G., Melegh B., Svennson L., Van Duijnhoven Y., Koopmans M. — (2004) Increase in viral gastroenteritis outbreaks in Europe and epidemic spread of new norovirus variant. Lancet, 363, (9410), 682-688.
[14] Kamel A.H., Ali M.A., El-Nady H.G., De Rougemont A., Pothier P., Belliot G. — (2009) Predominance and circulation of enteric viruses in the region of Greater Cairo, Egypt. J. Clin.
Microbiol., 47 (4), 1037-1045.
[15] Donaldson E.F., Lindesmith L.C., Lobue A.D., Baric R.S. — (2010) Viral shape-shifting:
norovirus evasion of the human immune system. Nat. Rev. Microbiol., 8, (3), 231-241.
Bull. Acad. Natle Méd., 2010, 194, no 8, 1427-1438, séance du 9 novembre 2010